番茄作为世界公认的模式植物并具有极高的营养价值,其育种历史可以主要分为驯化和改良两大阶段,而代谢物由于“搭车效应”在群体中展现出丰富的多样性。以往对植物群体代谢多样性的研究主要基于SNP这种遗传标记,但作为重要表观遗传修饰的DNA甲基化,特别是群体DNA甲基化与驯化改良的关系以及对代谢多样性的影响却尚未可知。

2023年03月23日,海南大学三亚南繁研究院/热带作物学院王守创课题组在国际学术期刊Science China Life Sciences(中科院1区,TOP)上发表题目为“Population analysis reveals the roles of DNA methylation in tomato domestication and metabolic diversity”的研究论文。该研究对近百个番茄品种的叶片进行全基因组甲基化测序,产生了约100亿对的双端测序数据,共鉴定了8375个DMR(Different methylation region),绘制了番茄首个群体级别的表观遗传变异图谱。随后整合变异组、转录组和代谢组等多组学进行分析,得到了超过3万个基因的群体表达矩阵,鉴定了339种代谢物,挖掘了数千个与代谢物显著关联的变异位点。该研究解析了番茄群体代谢多样性与育种过程中DNA甲基化变异的关系,构建了多组学关联网络并完善了番茄多酚等代谢物的合成通路。这项研究不仅为番茄遗传改良和新品种培育提供了重要的源头数据,也为深刻认识植物代谢多样性的分子和遗传基础提供了理论参考。
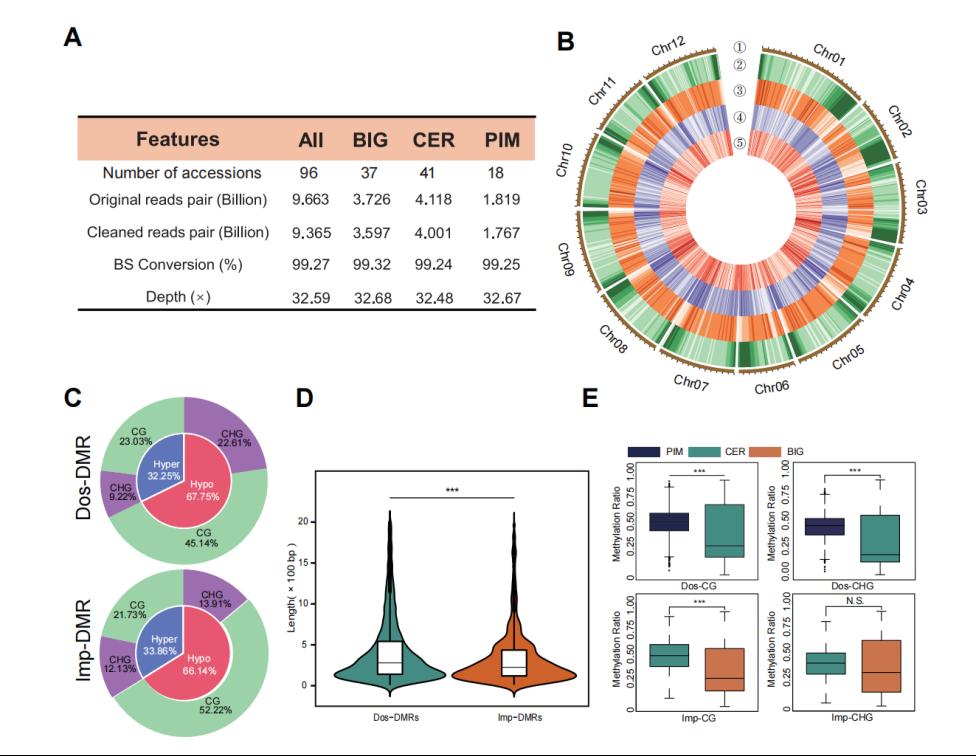
图1. 驯化和改良过程中DNA甲基化变异总结。A全基因组亚硫酸氢盐测序数据概括。B DMR在番茄染色体上的分布。C 番茄驯化改良过程中DMR分类。D 不同育种过程和序列背景DMR长度比较。E 驯化和改良过程中DMR信号比较。
研究者基于WGBS(Whole Genome Bisulfite Seuqneicng)测序技术分别在驯化和改良过程中鉴定到6801和1574个DMRs,并且它们广泛分布于注释为基因的区域,在这两个过程中Hypo-DMRs都占较大比例。其后通过DMRs的比较分析,发现在番茄育种历史中DMRs的数目、长度和信号水平都是逐步降低的(图1)。这些结果表明在番茄育种历史过程中,群体DNA甲基化在多个维度上发生了巨大变异。
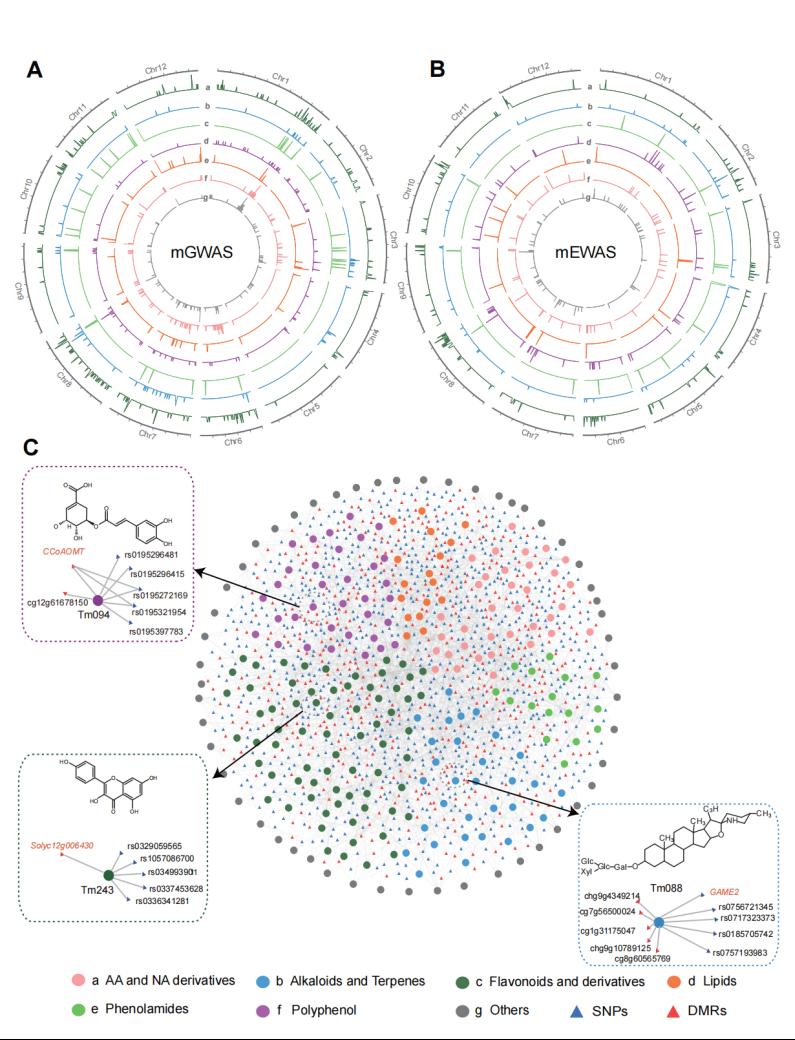
图2. 多组学关联网络整合与构建。A和B mEWAS和mGWAS定位结果分布。C 基于代谢物、SNPs和DMRs关联分析的多组学网络。
研究者进一步通过非靶向代谢组学的方法在番茄叶片中一共鉴定到339个高质量代谢物,利用mGWAS(metabolite-based Genome-Wide Association Study)和mEWAS(metabolite-based Epigenetic-Wide Association Study)分别鉴定到了971和711个显著关联的大效应位点,绘制了代谢物-单核苷酸多态性-差异甲基化区域的多组学关联网络,并挖掘到多个调控代谢物生物合成的候选基因(图2)。基于这些结果,研究者表明一些候选基因受到遗传和表观遗传变异组合的影响,而另一些则只受到一种类型的变异作用。
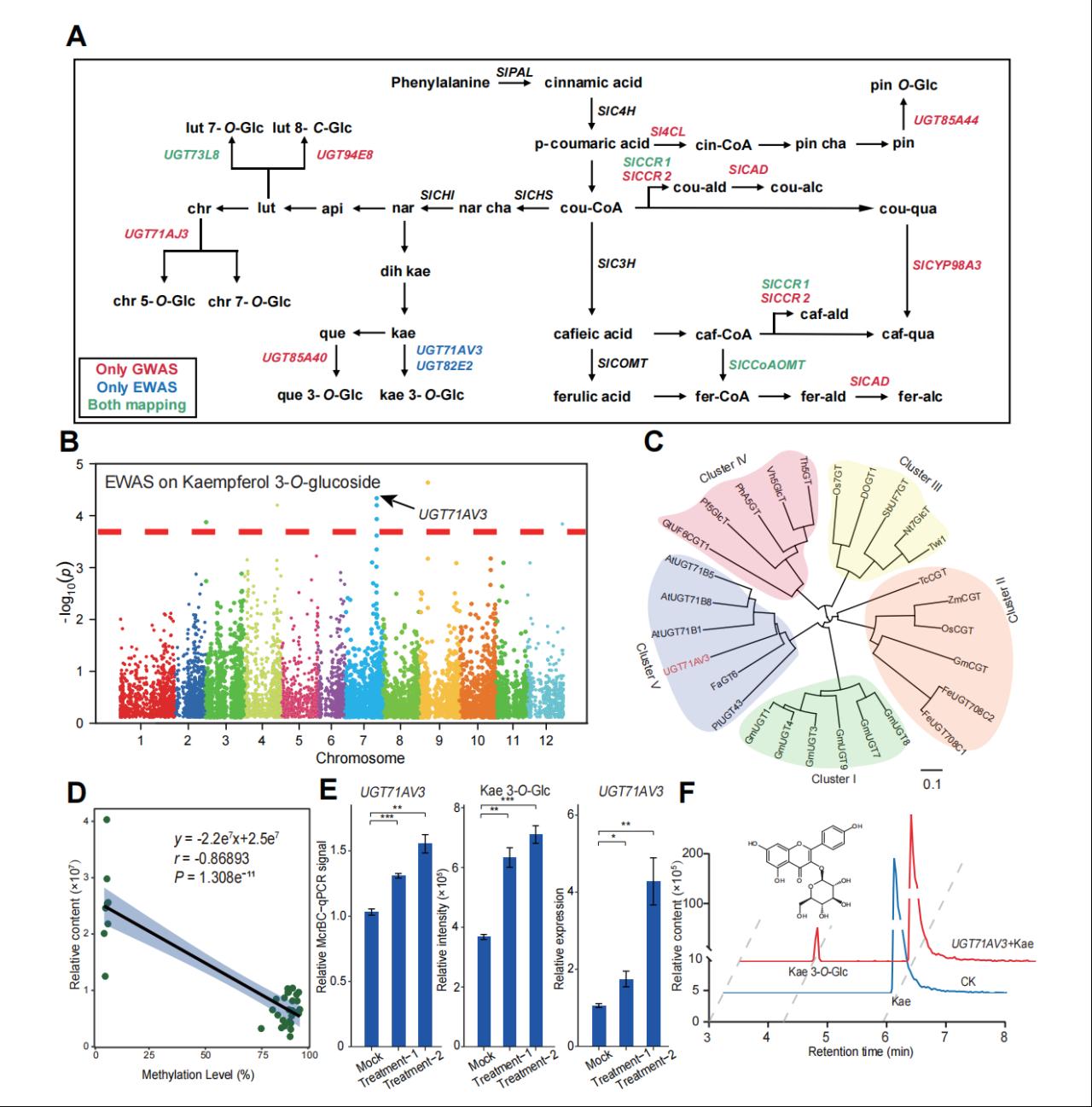
图3. 基于mGWAS和mEWAS绘制的番茄多酚生物合成途径。A 显示了在多酚生物合成途径中所涉及的已知基因和本研究鉴定的候选基因;B kaempferol 3-O-glucoside的EWAS曼哈顿图。C 类黄酮糖基转移酶基因的系统发育树。 D DMR (chg7g56698981)的甲基化水平与kaempferol 3-O-glucoside代谢物水平的相关性分析。E H2O和5-Aza处理后叶中相对DNA甲基化水平、UGT71AV3相对表达量和kaempferol 3-O-glucoside相对含量的展示。F, UGT71AV3和kaempferol作为底物的体外活性色谱图。
研究者基于多组学关联网络在番茄多酚合成通路中一共鉴定到13个候选基因。通过mEWAS发现七号染色体上的UGT71AV3与kaempferol 3-O-glucoside含量显著关联,而在mGWAS中并没有显著关联的位点。利用系统发育树和相关性分析初步鉴定UGT71AV3为kaempferol三号位氧糖基化修饰的候选基因。随后,采用5-Aza处理和体外实验表明,DNA甲基化水平被抑制后UGT71AV3的转录水平提高,从而导致kaempferol 3-O-glucoside的积累量增多,证实了UGT71AV3具有催化kaempferol三号位氧糖基化修饰的功能(图3)。总的来说,这些结果表明DMR可以和SNP共同影响番茄代谢物的生物合成,同时也可以鉴别到SNP无法捕获的候选基因,丰富了对代谢多样性的见解。
海南大学博士研究生郭昊,曹鹏,硕士研究生汪超,赖军和邓渊为该论文的共同第一作者。海南大学三亚南繁研究院/热带作物学院王守创教授为该论文的通讯作者。海南大学杨君副教授,德国马克思-普朗克研究所Fernie教授对本研究给予了重要的指导建议。本研究获得海南省自然科学基金重点研发项目、国家自然科学基金、科技部重点研发项目、中国科协青年人才托举工程、海南省院士创新平台项目,海南大学启动基金的支持。
论文信息:https://doi.org/10.1007/s11427-022-2299-5
 海南大学
海南大学 海南大学
海南大学