近日,海南大学药学院罗海彬教授团队在生物信息学领域国际权威期刊《Briefings in Bioinformatics》(IF因子: 10.5; 中国科学院一区)在线发表题为“3D-SMGE: A Pipeline for Scaffolds-based Molecular Generation and Evaluation”的研究论文,该研究发展了药物设计新方法3D-SMGE,可作为人工智能辅助药物分子生成的神经网络管道,包含高效的3D分子生成模型和高精度的ADMET性质预测,为创新药物的结构优化提供了一种具有前景的药物设计新方法。该论文还以创新药物重要靶标PDE4为例,发现一系列具有良好ADMET性质与基础性质的类药性分子。
3D-SMGE工作流程,从数据处理、模型训练、基于骨架的分子生成到性质评估。
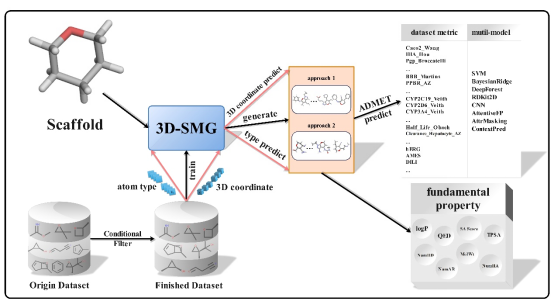
在药物发现的过程中,关键问题之一就是如何从特定结构出发通过结构改造获得更佳生物活性和ADMET性质,也称为结构优化。基于一个起始的核心骨架,使用深度生成模型生成具有所需性质的类药分子,将为加速结构优化过程提供强大工具。为了有效地从特定骨架生成分子,并为结构优化提供基础,该论文提出了3D-SMGE工作,包括分子生成模块与ADMET性质预测模块。对于分子生成模块,论文中使用基于原子类型与空间坐标的3D分子表征方式,提出了用于端到端设计的深度生成模型3D-SMG。在3D-SMG模型中,设计了交叉聚合连续滤波卷积(ca-cfconv),用于实现高效低成本的3D空间特征提取,同时确保了原子空间旋转与平移的不变性。3D-SMG也被证明能够生成有效、独特和新颖的类药分子。此外,论文中提出数据自适应多模型(data-adaptive multi-model)的ADMET预测方法在27个ADMET基准数据集中,其中24个数据集表现优于或保持最佳评估指标结果。
该研究成果以海南大学为第一单位,药学院硕士生许超为第一作者,药学院罗海彬教授、中山大学李哲副教授为共同通讯作者,药学院黄姝姮副教授等参与课题研究和指导。研究工作得到了国家自然科学基金、海南省自然科学基金等项目支持。
全文链接:https://academic.oup.com/bib/article-abstract/24/6/bbad327/7281738
 公安部备案号:46010802000190Copyright © 2024 开云app体育
公安部备案号:46010802000190Copyright © 2024 开云app体育




